Amazon Web Services ブログ
Amazon SageMakerによるマルチモーダルなヘルスデータの機械学習モデルトレーニング
この記事は、”Training Machine Learning Models on Multimodal Health Data with Amazon SageMaker” を翻訳したものです。
これは、マルチモーダル機械学習(マルチモーダルML)に関する2部構成のブログ記事です。第1部では、RNA配列データ、臨床データ(EHRデータからの引用)、およびアノテーションを含む医用画像を処理するためのパイプラインを実装しました。この記事では、各データモダリティから特徴量をプールし、非小細胞肺がん(NSCLC)と診断された患者の生存を予測するモデルを学習する方法を説明します。最初のブログ記事に基づいて、RNA配列データ、臨床データ、医用画像、およびそれらの画像のアノテーションで構成されるNSCLC Radiogenomicsデータセットを引き続き使用していきます[1]。
マルチモーダルMLモデルは、個別治療、臨床診断支援、薬物応答予測など、他のアプリケーションに適用できます。また、ゲノム、臨床、医用画像への応用を紹介しますが、アプローチとアーキテクチャは、幅広いヘルスデータMLユースケースとフレームワークに適用できます。さらに、このアプローチは、バッチおよびリアルタイム推論のユースケースの学習モデルに適用できます。
ウォークスルー
このシリーズの第1部では、図1に示すように、マルチモーダルデータパイプラインのアーキテクチャを紹介しました。ゲノム、臨床、および医用画像データを処理し、各モダリティから処理された特徴量をAmazon SageMaker Feature Storeにロードしました。この記事では、異なるモダリティからの特徴量を取り出し、1つまたは2つのデータモダリティで学習したものを上回る予測モデルを学習する方法を説明します。
図1: マルチモーダルヘルスデータの統合と分析のためのアーキテクチャ
次に、各データモダリティから取り出された特徴量は、特徴量を格納するための専用リポジトリであるAmazon SageMaker Feature Storeに書き込まれます。特徴量準備とモデル学習はAmazon SageMaker Studioを使用して実行されます。
前提条件
このシリーズの第1部を完了した場合は、前提条件とSageMaker Studioの利用手順をスキップして、以下のマルチモーダルFeature Storeに進むことができます。
このウォークスルーの前提条件は次のとおりです。
- Amazon SageMaker、Amazon S3、Amazon Athenaをプロビジョニングする権限を持つAWSアカウント。
- このブログに付属するコードリポジトリへのアクセス。
このブログ記事で紹介された分析を実行すると、SageMakerの無料利用枠内に収まるか、あるいは5ドル未満になります。
ステップセクションの作成
SageMaker Studio
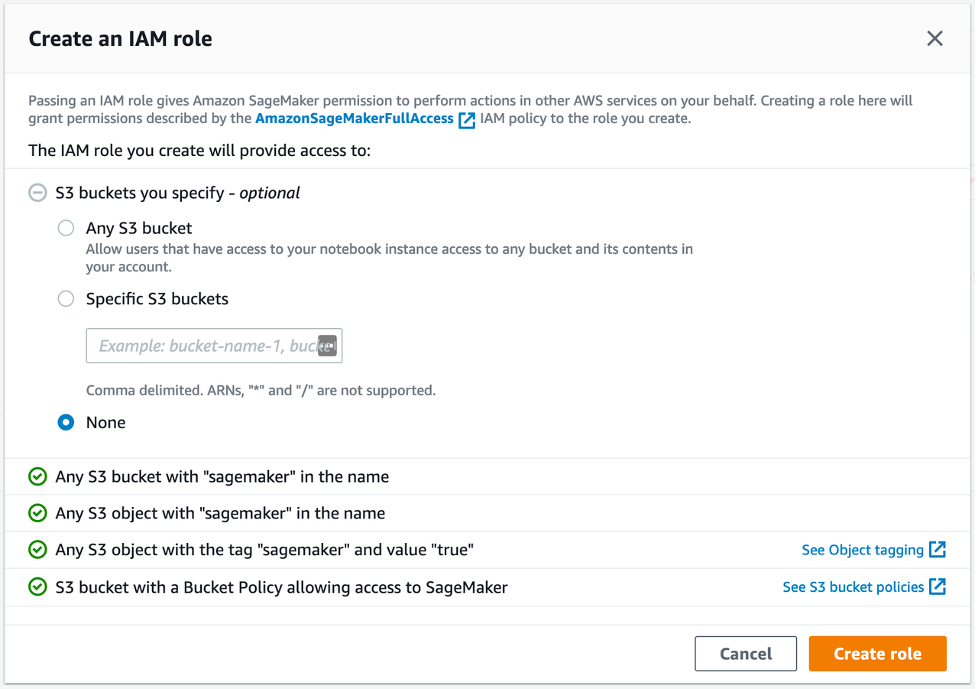
SageMaker Studioを使用して、データの処理、Jupyter Notebookの利用、およびゲノミクスのパイプラインで使用されるEC2インスタンスへのアクセスが可能です。開始するには、Identity and Access Management (IAM) を使用した標準セットアップの手順に従って、アカウントのSageMaker Studioにアクセスします。簡単のため、図2に示すように、Execution roleのCreate roleを選択し、S3バケットを明示的に指定しないでください。タグ「sagemaker」と値「true」を使用して、SageMakerのS3オブジェクトへのアクセスを許可します。この方法では、S3の入力と出力データのみにアクセスできます。
図2: SageMakerのIAMロールの設定
ネットワークのセクションで、VPCでのSageMaker Studioへのアクセスを選択し、プライベートサブネットを指定します。また、AppNetworkAccessTypeを VpcOnlyに設定して、インターネットからの直接アクセスを無効にします。
[送信]を選択してstudioを作成し、環境がプロビジョニングされるまでしばらく待ちます。SageMaker Studio IDEが使用可能になったら、左側のツールバーでGitアイコンを選択し、このブログに付属するリポジトリを複製します。
デフォルトでは、SageMaker Studioのノートブック、ローカルデータ、およびモデルアーティファクトはすべてCustomer Master Key (CMK)で暗号化されます。この例では、匿名化されたデータを扱っています。ただし、Protected Health Information(PHI)を使用する場合、保存中および転送中のすべてのデータを暗号化し、狭義に定義された役割を適用し、最小権限の原則に従ってアクセスを制限することが重要です。ベストプラクティスに関する詳しいガイダンスについては、ホワイトペーパー「HIPAA セキュリティとコンプライアンスの設計」を参照してください。
マルチモーダルFeature Store
SageMaker Feature Storeを使用して、マルチモーダルMLモデルを学習するために、さまざまなデータソースから取得した特徴量を格納および管理します。FeatureGroupには、Feature Storeに格納されているすべてのデータのメタデータが含まれます。SageMaker Feature Storeでは、オンラインストアとオフラインストアの2種類のストアがサポートされています。オンラインストアは GetRecord APIを提供して、低レイテンシーでリアルタイムでレコードの最新の値にアクセスできます。一方、オフラインストアはすべてのバージョンの機能を保持し、学習データを作成するときのバッチアクセスに最適です。ゲノム、臨床、医用画像など、データタイプごとに特徴量グループを作成し、以前に構築された特徴量ベクトルを対応する特徴量グループのレコードとして、それぞれの処理パイプラインの一部として追加します。次のコードスニペットは、S3バケットからゲノム二次解析パイプラインによって生成された出力データを取得し、このデータに固有のFeatureGroupを作成し、それを格納するために取り込む方法を示しています。
実装の詳細は、ゲノム、臨床、画像フォルダ内のpreprocess-genomic-data.ipynb、preprocess-clinical-data.ipynb、preprocess-imaging-data.ipynbスクリプトにあります。
data_gen = pd.read_csv('s3://{}/{}'.format(<S3-BUCKET>, <FILE-NAME>))
feature_store_session = Session(
boto_session=<BOTO3-SESSION>,
sagemaker_client=<BOTO3-SESSION-CLIENT-FOR-SAGEMAKER>,
sagemaker_featurestore_runtime_client=<BOTO3-SESSION-CLIENT-FOR-SAGEMAKER-FEATURESTORE-RUNTIME>
)
genomic_feature_group_name = 'genomic-feature-group-' + strftime('%d-%H-%M-%S', gmtime())
genomic_feature_group = FeatureGroup(name=genomic_feature_group_name, sagemaker_session=feature_store_session)
genomic_feature_group.create(
s3_uri=f"<S3-DEFAULT-BUCKET>",
record_identifier_name=<ID>,
event_time_feature_name=<EVENT-TIME>,
role_arn=<ROLE>,
enable_online_store=True)
genomic_feature_group.ingest(data_frame=data_gen, max_workers=3, wait=True)
MLトレーニングの場合は、3つの特徴量グループに対してクエリを実行して、オフラインストアに格納されているデータを結合します。与えられたデータセットについて、この積算により119のデータサンプルが得られ、各サンプルは 215次元のベクトルになります。
genomic_table = <GENOMIC-TABLE-NAME>
clinical_table = <CLINICAL-TABLE-NAME>
imaging_table = <IMAGING-TABLE-NAME>
query = <FEATURE-GROUP-NAME>.athena_query()
query_string = 'SELECT '+genomic_table+'.*, '+clinical_table+'.*, '+imaging_table+'.* \
FROM '+genomic_table+' LEFT OUTER JOIN '+clinical_table+' ON '+clinical_table+'.case_id = '+genomic_table+'.case_id \
LEFT OUTER JOIN '+imaging_table+' ON '+clinical_table+'.case_id = '+imaging_table+'.subject \
ORDER BY '+clinical_table+'.case_id ASC;'
query.run(query_string=query_string, output_location='s3://'+<BUCKET>+'/'+prefix+'/query_results/')
imaging_query.wait()
multimodal_features = query.as_dataframe()
実装の詳細は、コードリポジトリのmodel-train-testフォルダ内のtrain-test-model.ipynbの「Get features from SageMaker FeatureStore based on data type」の箇所を参照してください。
モデルトレーニング
デモンストレーションの目的で、患者の生存を予測するための分類タスクを検討します。MLモデルをトレーニングするには、SageMaker XGBoost コンテナを使用して勾配ブースティングライブラリXGBoostのエスティメーターを構築します。上記の手順で作成した結合データセットを使用して、さらに分析します。このデータをランダムにシャッフルし、学習に80%、テストに20%として分割します。さらに、学習のデータから学習に80%、モデルの検証に20%として分割します。特徴量のスケーリングを使用して、独立した特徴量の範囲を正規化します。最も固有な特徴量を特定するには、統合特徴量に対して主成分分析(PCA) を実行します。データの99%の分散に寄与する上位の主成分を特定します。具体的には、これにより、215の特徴から65の主成分に次元が減少し、教師あり学習の特徴量を構成します。現在の目的は、マルチモーダルデータを使用してベースラインモデルを学習することであるため、デフォルトのハイパーパラメーターを使って、ハイパーパラメーターの調整は実行しません。他の分類アルゴリズムを採用し、ハイパーパラメーターの調整を実施すると、結果が改善される可能性があります。
マルチモーダルMLモデル学習の実装詳細は、コードリポジトリのmodel-train-testフォルダ内のtrain-test-model.ipynbの[Split data for training and testing]セクションと[Train model]セクションの間にあります。
モデル評価
ゲノム、臨床、および医用画像データモダリティだけで学習された場合の生存結果を予測するモデルの性能を最初に評価します。次に、すべてのデータドメインを結合した特徴量、つまりマルチモーダルデータで学習したモデルの性能を評価します。比較分析では、それぞれの精度、F1スコア、適合率、および再現率を計算します。表1に示すように、マルチモーダルデータは、単一のドメインのデータを使用するよりも高い予測性能につながります。この結果は、4つの評価指標すべてで一貫しています。対応するコードは、コードリポジトリのmodel-train-testフォルダ内のtrain-test-model.ipynbの[Test model]セクションにあります。
表1: 生存結果の予測モデル学習において、異なるデータドメインを使用した評価指標の比較
この結果をよりよく理解するために、特定の予測タスクにおける各データドメインの重要性をさらに調べてみましょう。異なるモダリティに属するデータの重要度または判別特性を判断するには、特徴量として使用された主成分の構成を調べます。これらの主成分は、各モダリティから取得した元の特徴量の線形組み合わせです。これらは、データ内の分散の99%を表す直線的に相関のない特徴量のセットを構成します。最初の主成分は最大の分散を説明し、後続の主成分はそれぞれデータ内の分散を連続的に少なく説明しています。図3は、上位20の主成分について、固有性によって色分けされた元の特徴量によって説明される分散を示します。最初の数個の成分については、医用画像の特徴量は、臨床およびゲノム特徴よりも重み付けが高く、分散が大きく(図3のヒートマップでは強度が高い)ため、生存結果予測における医用画像の特徴量の重要性が示されている。しかし、第3主成分から、臨床およびゲノムモダリティの重み付けが見られます。これは、臨床およびゲノムモダリティが、医用画像から得られるものを超える追加の変数が寄与していることを示しています。図4の可変相関円にも同じ傾向が見られます。第1および第2の主成分に寄与する上位10の特徴量は、医用画像領域からのものです。しかし、図4に示すように、ゲノム特徴量(gdf_15など)および臨床特徴量(喫煙期間など)は、第4および第5主成分に寄与しています。
ヒートマップと相関円を生成するコードは、コードリポジトリのmodel-train-testフォルダ内のtrain-test-model.ipynbの[Get feature importance]セクションおよび[Plot Correlation Circle]セクションにあります。
図3: 上位20の主成分について、ドメインごとに色分けされた各特徴量によって説明される変数を示すヒートマップ。X軸は主成分を表し、Y軸は異なるドメイン(ゲノム、臨床、医用画像)から得られた特徴量を示し、セルの濃度は対応する主成分に対するその特徴量の寄与レベル(正規化値)を示します。最初の数個の主要成分については、医用画像の特徴量は高い寄与を持ち、続いて臨床およびゲノムの特徴量が続きます。これは、特定のデータセットについて、医用画像から得られた特徴量が、生存転帰を予測する上で最も特徴的であるか、または重要であることを示している。ただし、すべてのモダリティには、モデルのパフォーマンスを向上させることができる情報が含まれている。
図4: 第4主成分と5番目の主成分の上位25の特徴量または変数間の相関を示す変数相関円。一対のベクトル間の角度は、ベクトル間の相関レベルを示します。小さい角度は正の相関を示し、180度に近い角度は負の相関を示します。特徴量と原点との間の距離は、プロットで特徴量がどれだけうまく表されているかを示します。これらの主成分について、ゲノム(例:gdf15)、臨床(例:pack_years、histology_adinocarcinoma)、および医用画像(例:original_glcm_clustershade)の3つのドメインから上位25の特徴量が得られる。
環境削除
今後の料金が発生しないようにするには、上記の手順で作成したリソースを削除します。
- SageMaker Studioのファイルメニューから、[Shutdown All]を選択し、SageMakerダッシュボードの[Recent Activity]セクションに表示されているリソースを削除します。
- Athenaコンソールから、この解析で作成したテーブルをすべて削除します。
- S3に保存されているすべてのデータを削除します。
まとめ
この2部構成のブログシリーズでは、一連のデータ分析パイプラインとMLツールを実装して、多様な非構造化データモダリティからデータを効率的に処理する方法を説明しました。フルマネージド型のAWSサービスを活用することで、セットアップを合理化し、マルチモーダルデータを大規模に処理し、インフラストラクチャではなくMLモデリングに集中しやすくしました。リアルワールドデータに関する実験的評価を通じて、複数のモダリティからデータを統合することで、モデルの予測能力を高めることを実証しました。
マルチモーダルデータを活用することで、ヘルスケアおよびライフサイエンス向けのより良いMLモデルが得られ、その後のケア提供と患者の転帰が改善されます。ゲノミクス、臨床データ、医用画像に重点を置いていますが、このアプローチは他のデータモダリティにも適用できます。この例を、最も関心のあるマルチモーダルデータアプリケーションに拡張することをお勧めします。
AWSのヘルスケアとライフサイエンスに関する詳細については、thinkwithwp.com/healthをご覧ください。
参考資料
[1] Bakr, Shaimaa, et al. “A radiogenomic dataset of non-small cell lung cancer.” Scientific data 5.1 (2018): 1-9.
著者について
Olivia Choudhury
Olivia Choudhury, PhDはAWSのパートナーソリューションアーキテクトです。彼女は、ヘルスケアおよびライフサイエンス分野のパートナーの、AWS を活用した最先端ソリューションの設計、開発、拡張を支援しています。彼女は、ゲノミクス、ヘルスケア分析、連合学習、プライバシー保護機械学習のバックグラウンドを持っています。仕事以外では、ボードゲームをしたり、風景を描き、漫画を集めています。
Andy Schuetz
Andy Schuetz, PhDはAWSのシニアスタートアップソリューションアーキテクトで、お客様がヘルスケアおよびライフサイエンスソリューションを利用できるよう支援することに注力しています。AndyがAWSで構築作業をしていないとき、彼はマウンテンバイクに乗ることが好きです。
Michael Hsieh
Michael Hsiehは、シニアAI/MLスペシャリストソリューションアーキテクトです。彼はHCLSのお客様と協力して、AWSテクノロジーと医用画像処理に関する専門知識を活用してMLジャーニーを進めています。シアトルの住民として、彼はハイキングコース、SLUでのカヤック、シルスホール湾の夕日など、街が提供する偉大な母なる自然を探索するのが大好きです。
翻訳はIndustry Solutions Architectの松永が担当しました。原文はこちらです。